코드링크
비최대 억제(Non-Maximum Suppression)
이전에 포스팅한 모라벡, 해리스, 헤시안 행렬, 슈산 모두 어떤 점에 대해 특징일 가능성을 측정해주었다.
하지만 코너에서 한 점만 큰 값을 갖는 것이 아니라 큰 값이 일정한 범위에 퍼져있어 한 지점을 선택하는 방법이 필요하며 이러한 일을 위치 찾기(Localization)이라 부른다(SLAM 분야의 localization과는 다르다). 가장 합리적인 방법으로 지역 최대점을 취하는 것인데, 이름에서 알 수 있듯이 지역내에서 최대가 아니면 억제되고 지역내에서 최대라면 특징점으로 결정된다.
일반적으로 지역을 정할 때는 동서남북의 네 이웃 화소만 보는 4-연결 방식과 대각선을 포함한 8-연결 방식이 있다. 물론 상황에 따라 더 넓은 지역을 이웃으로 취급할 수도 있다.
지역내에서 최대값이더라도 특징점으로 보기 애매한 경우가 있다. 이를 위해 지역내에서 최대값이더라도 임계값을 설정하여 임계값보다 작은 경우 잡음(noise)로 취급한다.
brown은 특징점이 영상의 특정 부분에는 밀집되어 있고 다른 부분은 드물게 분포하는 문제를 해결하기 위해 특징점에 대해 지역 내에서 최대이며 주위 화소보다 일정 비율 이상 커야 한다는 조건을 만들었다. 이를 적응적 비최대 억제 방법(adaptive non-maximum suppression)이라고 한다.
코드 구현
이번 구현에서는 이전에 구현했던 harris corner를 이용해 특징점의 수가 얼마나 주는 지 확인 할 것이다.
from util import *
import matplotlib.pyplot as plt
import numpy as np
import cv2
img = cv2.imread('./data/red_deer.jpg',cv2.IMREAD_GRAYSCALE)
plt.imshow(img,cmap='gray')
plt.show()
우선 지역이 4-연결 구성일 때와 8-연결 구성일 때에 대해 구현하였으며 threshold는 임의로 지정하였다. 사실 이 구현에서는 harris를 검출할 때 임계값을 설정 했었기 때문에 이웃보다 크기만 하면 검출된다.
def NMS(feature,n=4,threshold=0.02):
feature = np.expand_dims(feature,-1)
# 상하좌우
n_r = np.pad(feature[1:], ((0, 1), (0, 0),(0,0)))
n_l = np.pad(feature[:-1], ((1, 0), (0, 0),(0,0)))
n_d = np.pad(feature[:,1:], ((0, 0), (0, 1),(0,0)))
n_u = np.pad(feature[:,:-1], ((0, 0), (1, 0),(0,0)))
if n==8: # 대각선
n_ul = np.pad(n_u[:-1], ((1, 0), (0, 0),(0,0)))
n_ur = np.pad(n_u[1:], ((0, 1), (0, 0),(0,0)))
n_dl = np.pad(n_d[:-1], ((1, 0), (0, 0),(0,0)))
n_dr = np.pad(n_d[1:], ((0, 1), (0, 0),(0,0)))
ret = np.concatenate([feature,n_r,n_l,n_d,n_u,n_ul,n_ur,n_dl,n_dr],axis=-1)
else:
ret = np.concatenate([feature,n_r,n_l,n_d,n_u],axis=-1)
ret = np.expand_dims(np.argmax(ret,-1),-1) # 최대값을 가지는 index
return np.squeeze(np.where(np.logical_and(ret==0,feature>threshold),feature,0)) # ret이 0 이라는 것은 지역보다 크다는 것을 뜻한다.
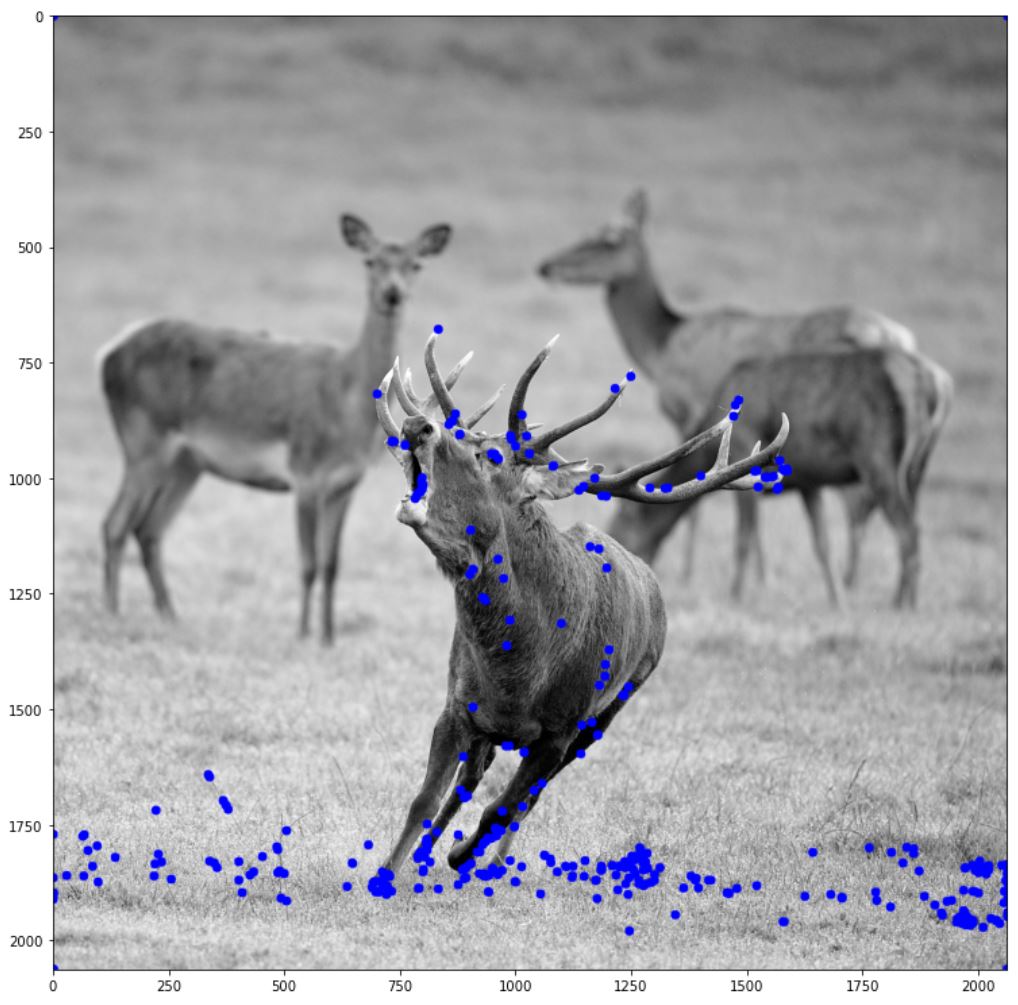
우선 NMS 적용전과 적용후의 그림이다.
harris = Harris_corner(img,threshold=0.02)
fig = plt.figure(figsize=(13,13))
plt.subplot(121)
plt.imshow(draw_featrue_point(img,harris,dot_size=5))
plt.xlabel('before NMS')
NMS_harris = NMS(harris)
plt.subplot(122)
plt.imshow(draw_featrue_point(img,NMS_harris,dot_size=5))
plt.xlabel('after NMS')
fig.tight_layout()
plt.show()
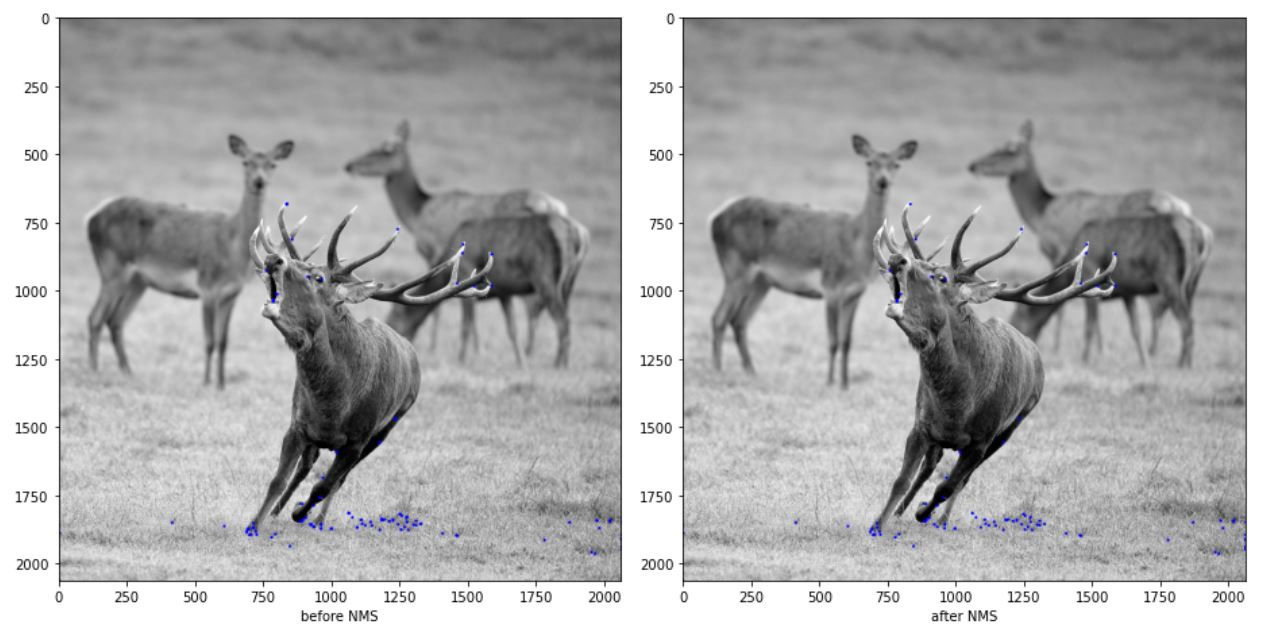
원의 굵기가 얇아진것 같기는 하지만 그림으로 봐서는 확실하게 줄어든지 잘 모르겠다. 확실하게 하기 위해 0보다 큰 값들을 카운팅해보자.
>>> print(np.sum(harris>0),np.sum(NMS_harris>0))
292 106
확실히 특징점의 갯수가 3배 가량 줄어 든 것을 확인할 수 있다.
코드링크
2차 미분을 사용한 방법(헤시안 행렬)
특징점을 구하는 또 다른 방법으로는 2차 미분을 사용한 방법이 있다.
이 방법에서는 아래의 헤시안 행렬(Hessian matrix)를 사용한다.
\[\mathbf{H} = \begin{pmatrix}d_{yy} & d_{yx} \\ d_{yx} & d_{xx} \end{pmatrix}\]
여기서 \(d_{yy}\)와 \(d_{xx}\)는 각각 y방향과 x방향으로 두 번 미분한 2차 도함수를 말하고 \(d_{yx}\)는 y방향으로 미분한 후 그 결과를 다시 x방향으로 미분한 도함수이다.
미분을 사용할 때의 주의점은 미분이 잡음을 증폭시킨다는 것이다. 심지어 2차 미분이므로 이를 바로 이미지에 적용하지 않는다. 대신 가우시안으로 스무딩을 거친 다음 그 결과에 2차 미분을 수행한다. 식으로 표현하면 다음과 같다.
\[\mathbf{H} = \begin{pmatrix}d_{yy}(\sigma) & d_{yx}(\sigma) \\ d_{yx}(\sigma) & d_{xx}(\sigma) \end{pmatrix} \\
where, d_{st}(\sigma) = \frac{\partial}{\partial t} \left( \frac{\partial}{\partial s}(G(y,x,\sigma) \circledast f(y,x))\right)\]
헤시안에서는 보통 두 종류의 값을 계산하는데 이들이 특징 가능성 값에 해당한다. 2번째 식은 LOG(Laplacian of Gaussian)는 에지 검출에 사용하였던 연산자와 같은 것이다. 이 연산자는 에지 근방에서 특징점이 많이 발생하므로, 에지를 걸러 주는 후처리가 따로 필요하다(이 방법은 나중에 다룬다).
\[C=det(\mathbf{H}) = d_{yy}(\sigma)d_{xx}(\sigma)-d_{yx}(\sigma)^2\]
\[C = \nabla^2 = trace(\mathbf{H}) = d_{yy}(\sigma) + d_{xx}(\sigma)\]
슈산(SUSAN)
SUSAN(Smallest Univalue Segment Assimilating Nucleus) 방식은 현재 처리 중인 중심점과 인근 지역의 밝기 값이 얼마나 유사한가를 따져 보고 그 결과에 따라 특징일 가능성을 측정한다.
SUSAN에서는 중심점에 원형 마스크를 씌우고 중심점과 명암값이 유사한 점으로 구성된 영역을 USAN(Univalue Segment Assimilating Nucleus)라 부른다. SUSAN은 마스크와 우산의 크기 비율을 측정한 후, 50% 정도인 곳을 에지, 50%보다 작은 곳을 코너로 검출한다.
결국 SUSAN 검출 알고리즘의 핵심은 아래의 식에 해당하는 우산의 크기를 측정해 주는 연산이다.
\[usan_area(r_{0}) = \sum_{r}(r,r_{0}) \\
where, s(r,r_{0}) = \begin{cases}1, \left| f(r)-f(r_{0}) \right| \le f_{1} \\ 0, Othere wise \end{cases}\]
여기서 usan_area는 중심점 r0에서 USAN의 크기이며 r은 원형 마스크 내의 화소들이다. 식에 따르면 usan_area(r0)는 명암값 차이가 t1 보다 작은 화소의 개수가 된다.
이제 USAN의 크기를 특징 가능성으로 표현해주기 위해 다음의 식을 사용한다.
\[C = \begin{cases}q-usan \ area(r_{0}),\quad usan \ area(r_{0}) \le f_{2} \\ 0, \qquad \qquad \qquad \quad Other wise \end{cases}\]
여기에서 t2는 보통 마스크 넓이의 50%로 설정하고, q는 0.75 x 마스크의 넓이로 설정한다.
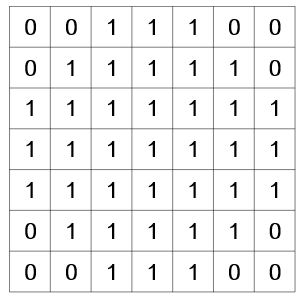
에서는 아래와 같은 7x7 크기, 넓이 37를 갖는 원형 마스크를 사용했다. 그리고 SUSAN을 이용하여 에지를 검출할 때는 t2를 마스크 넓이의 75%로 설정한다.
코드 구현
구현은 다음의 사진에 대해 진행한다.
import numpy as np
import matplotlib.pyplot as plt
import cv2
from util import *
img =cv2.imread('./data/red_deer.jpg',cv2.IMREAD_GRAYSCALE)
fig = plt.figure(figsize=(13,13))
plt.imshow(img,cmap='gray')
plt.show()

구현 자체는 단순하다. 그저 위의 식을 따라하면 된다. Gaussian은 이전에 구현한 것을 그대로 썼다(참고). dy와 dx는 이전에 Harris corner를 구현할 때 처럼 [-1,0,1] 마스크와 동일하게 동작하도록 구현했다. dy2와 dx2는 2차 미분에 해당하며 일차 미분한 이미지에서 동일한 작업을 한번 더 수행하면된다.
def Hessian_feature(img,threshold,sigma=1,kernel_size=3,max_value=255):
# Gaussian Smoothing
mask = get_gaussian_kernel(sigma,kernel_size)
img = (conv(img,mask)/max_value).squeeze()
# Second Derivative Y
dy = np.pad(img[1:], ((0, 1), (0, 0))) - np.pad(img[:-1], ((1, 0), (0, 0)))
dy2 = np.pad(dy[1:], ((0, 1), (0, 0))) - np.pad(dy[:-1], ((1, 0), (0, 0)))
# Second Derivative X
dx = np.pad(img[..., 1:], ((0, 0), (0, 1))) - np.pad(img[..., :-1], ((0, 0), (1, 0)))
dx2 = np.pad(dx[..., 1:], ((0, 0), (0, 1))) - np.pad(dx[..., :-1], ((0, 0), (1, 0)))
# Derivate Y and X
dydx = np.pad(dy[..., 1:], ((0, 0), (0, 1))) - np.pad(dy[..., :-1], ((0, 0), (1, 0)))
# Determinant of Hessian Matrix
Determinant = dy2*dx2 - (dydx**2)
# LOG (=Trace of Hessian Matrix)
LOG = dy2+dx2
return np.where(Determinant>threshold,Determinant,0), LOG
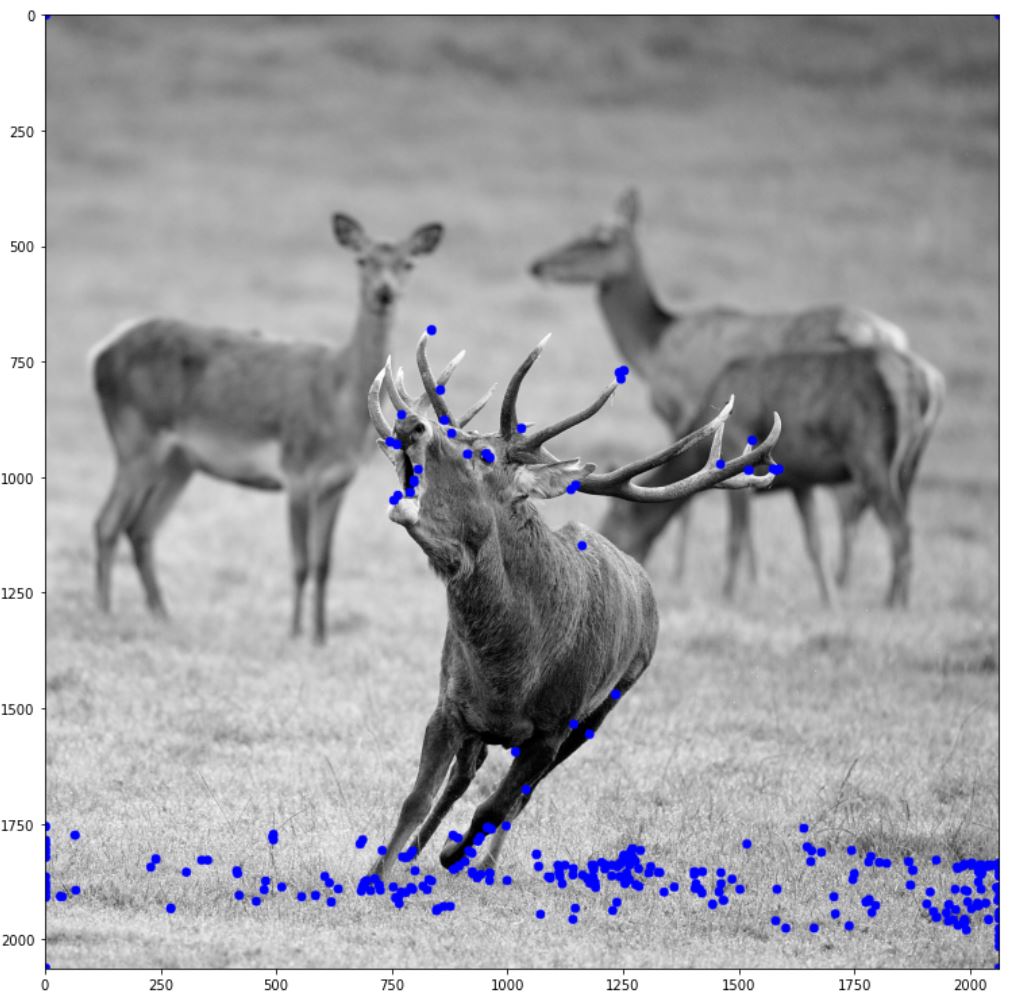
아래 그림은 0.1(threshold 값)을 경계로 검출된 특징점을 그린 그림이다. 그리는 함수는 이전에 Harris corner에서 구현한 “draw_harris_circle” 함수를 변경했다. 자세한 구현은 util.py를 참고하면 된다.
Det_C,LOG_C = Hessian_feature(img,0.1,1,7)
fig = plt.figure(figsize=(13,13))
plt.imshow(draw_featrue_point(img,Det_C))
plt.show()
다음의 그림은 이전에 구현한 LOG 연산자와 Hessian LOG를 비교한 그림이다. 중간 수식에서 값의 차이가 있기 때문에 완전히 동일하게 구현된 것은 아니지만 이론상 같은 내용인 것을 확인하기 위해 구현했다.
def Hessian_LOG_test(log_img,threshold=None):
if threshold == None:
threshold = np.max(log_img)*0.05
e_img = np.pad(log_img[:,1:],((0,0),(0,1)))
w_img = np.pad(log_img[:,:-1],((0,0),(1,0)))
n_img = np.pad(log_img[:-1,:],((1,0),(0,0)))
s_img = np.pad(log_img[1:, :], ((0,1), (0, 0)))
se_img = np.pad(s_img[:,1:],((0,0),(0,1)))
ne_img = np.pad(n_img[:, 1:], ((0, 0), (0, 1)))
sw_img = np.pad(s_img[:,:-1],((0,0),(1,0)))
nw_img = np.pad(n_img[:,:-1], ((0, 0), (1, 0)))
w_vs_e = np.int8(np.logical_and(np.absolute(w_img-e_img)>=threshold,sign(w_img,e_img)))
s_vs_n = np.int8(np.logical_and(np.absolute(n_img - s_img) >= threshold,sign(n_img,s_img)))
sw_vs_ne = np.int8(np.logical_and(np.absolute(sw_img - ne_img) >= threshold,sign(sw_img,ne_img)))
nw_vs_se = np.int8(np.logical_and(np.absolute(nw_img - se_img) >= threshold,sign(nw_img,se_img)))
return np.uint8(w_vs_e+s_vs_n+sw_vs_ne+nw_vs_se>=1)*255
fig = plt.figure(figsize=(13,13))
plt.subplot(121)
plt.imshow(Hessian_LOG_test(LOG_C),cmap='gray')
plt.xlabel("Hessian_LOG")
plt.subplot(122)
plt.imshow(get_LOG_img(img,1),cmap='gray')
plt.xlabel("LOG")
fig.tight_layout()
plt.show()

다음은 SUSAN 알고리즘을 구현할 것이다. 먼저 USAN 넓이를 구하는 함수는 다음과 같이 된다. 구현은 이전에 구현했던 im2col을 참고해 구현했다.
def usan_area(img, filter,threshold,max_value=255):
filter_h,filter_w = filter.shape
if len(img.shape) == 3:
img = cv2.cvtColor(img,cv2.COLOR_BGR2GRAY)
H, W = img.shape
img = np.expand_dims(img/max_value, -1)
u_pad_h = (filter_h-1)//2
d_pad_h = (filter_h-1)//2
l_pad_w = (filter_w-1)//2
r_pad_w = (filter_w-1)//2
if (filter_h-1) %2 ==1:
u_pad_h +=1
if (filter_w-1)%2 ==1:
l_pad_w +=1
input_data = cv2.copyMakeBorder(img, u_pad_h, d_pad_h, l_pad_w, r_pad_w, cv2.BORDER_CONSTANT)
input_data= np.expand_dims(input_data,-1)
input_data = np.transpose(input_data,(2,0,1))
col = np.zeros(( 1, filter_h, filter_w, H, W))
for y in range(filter_h):
y_max = y + H
for x in range(filter_w):
x_max = x + W
col[:, y, x, :, :] = input_data[:, y:y_max:1, x:x_max:1]
col = np.transpose(col,(0,3,4,1,2)).reshape(H*W, -1)
usan = np.abs(np.transpose(img,(2,0,1)).reshape([-1,1]) - col*filter.reshape((1,-1)))<=threshold
return np.sum(usan,axis=1).reshape(H,W)
SUSAN에서 사용한 원형 필터는 함수를 따로 작성했다. 사실 SUSAN에서 USAN_AREA에 대한 구현만 끝나면 나머지는 손쉽게 구현이 가능하다. 전체적인 함수는 아래와 같다.
def get_susan_filter():
filter = np.array([[0,0,1,1,1,0,0],
[0,1,1,1,1,1,0],
[1,1,1,1,1,1,1],
[1,1,1,1,1,1,1],
[1,1,1,1,1,1,1],
[0,1,1,1,1,1,0],
[0,0,1,1,1,0,0]])
return filter
def SUSAN(img,threshold,threshold2=None,q=None,filter=None):
if filter==None:
filter = get_susan_filter()
if threshold2 ==None:
threshold2 = np.sum(filter)*0.5
if q==None:
q = np.sum(filter)*0.75
USAN = usan_area(img,filter,threshold)
return np.where(USAN<=threshold2,q-USAN,0)
아래는 SUSAN을 이용해 특징점을 추출한 그림이다. threshold1 값은 임의로 0.4로 설정했다.
SUSAN_feat = SUSAN(img,0.4)
fig = plt.figure(figsize=(13,13))
plt.imshow(draw_featrue_point(img,SUSAN_feat),cmap='gray')
plt.show()
22 Jul 2020
•
Statistics
결합 분포
확률 변수 X,Y에 대해 \(W \equiv (X,Y)\)이 정의 될 때, W를 X,Y의 결합분포라고 부른다.
이산값(확률) VS 실수값(확률밀도)
| \(\ \ \ \ \ \ \ \\) |
이산값(확률) |
실수값(확률밀도) |
| 주변분포 |
\(P(X=a) = \sum_{y} P(X=a,Y=y)\) |
\(f_{X}(a) = \int^{\infty}_{-\infty}f_{X,Y}(a,y)dy\) |
| 조건부분포 |
\(P(Y=b \mid X=a) \equiv \frac{P(X=a,Y=b)}{P(X=a)} \\ P(X=a,Y=b) = P(Y=b \mid X=a) P(X=a)\) |
\(f_{Y \mid X}(b \mid a) \equiv \frac{f_{X,Y}(a,b)}{f_{X}(a)} \\ f_{X,Y}(a,b) = f_{Y \mid X} (b \mid a)f_{X}(a)\) |
| 베이즈 공식 |
\(P(X=a \mid Y=b) = \frac{P(Y=b \mid X=a)P(X=a)}{\sum_{x}P(Y=b \mid X=x)P(X=x)}\) |
\(f_{X \mid Y} (a \mid b) = \frac{f_{X \mid Y}(b \mid a)f_{X}(a)}{\int_{-\infty}^{\infty}f_{X \mid Y}(b \mid x)f_{X}(x)dx}\) |
| 독립성의 다른 표현 |
1. \(P(Y=b \mid X=a)\)가 \(a\)와 상관없다.
2. \(P(Y=b \mid X=a) = P(Y=b)\)
3. \(P(X=a, Y = 여러 가지)\)의 비가 \(a\)에 관계없이 일정하다.
4. \(P(X=a, Y=b) = P(X=a)P(Y=b)\)
5. \(P(X=a,Y=b) = g(a)h(b)\)의 형태 |
1. \(f_{Y \mid X} (b \mid a)\)가 \(a\)와 상관없다.
2. \(f_{Y \mid X} (b \mid a) = f_{Y}(b)\)
3. \(f_{X,Y} (a, 여러가지)\)의 비가 \(a\)에 관계없이 일정하다.
4. \(f_{X,Y}(a,b) = f_{X}(a)f_{Y}(b)\)
5. \(f_{X,Y}(a,b) = g(a)h(b)\)의 형태 |
| 기댓값 |
1. \(E[X] \equiv X(w) 그래프의 \ 부피\)
2. \(E[X] = \sum_{x} xP(X=x)\)
3. \(E[g(X)] = \sum_{x}g(x)P(X=x)\)
4. \(E[h(X,Y)] = \sum_{y} \sum_{x} h(x,y) P(X=x,Y=y)\)
5. \(E[aX+b] = aE[X] +b\) |
1. 동일
2. \(E[X] = \int^{\infty}_{-\infty} xf_{X}(x)dx\)
3. \(E[g(X)] = \int^{\infty}_{-\infty} g(x)f_{X}(x)dx\)
4. \(E[h(X,Y)] = \int^{\infty}_{-\infty}h(x,y)f_{X,Y}(x,y)dxdy\)
|
| 분산 |
1. \(V[X] \equiv E[(X-\mu)^2], \ \ \mu \equiv E[X]\)
2. \(V[aX+b]=a^2 V[X]\)
|
1. 동일
2. 동일 |
| 표준편차 |
1. \(\sigma_{X} \equiv \sqrt{V[X]}\)
2. \(\sigma_{aX+b} = \begin{vmatrix}a\end{vmatrix} \sigma_{X}\)
|
1. 동일
2. 동일
|
| 조건부 기댓값 |
1. \(E[Y \mid X = a] \equiv \sum_{b}bP(Y=b \mid X=a)\) |
1. \(E[Y \mid X =a] \equiv \int^{\infty}_{-\infty} yf_{Y}(y \mid X=a) dy\) |
| 조건부 분산 |
1. \(V[Y \mid X =a] \equiv E[(Y-\mu(a))^2 \mid X=a]\) |
동일 |
확률변수의 독립성
이산값과 유사하다.
\[f_{Y \mid X}(b \mid a) = f_{Y}(b)\]
가 항상 어떠한 a와 b에서도 성립할 때, X와 Y는 독립이다. 또한 아래도 성립한다.
\[f_{Y \mid X}(b \mid a) = \frac{f_{X,Y}(a,b)}{f_{X}(a)} \\
\Rightarrow f_{X,Y}(a,b) = f_{X}(a)f_{Y}(b) \\\]
결합분포의 변수변환
확률밀도함수의 변환 공식은 다음과 같다.
\[f_{Z,W}(z,w) = \frac{1}{\begin{vmatrix} \partial (z,w) / \partial (x,y)\end{vmatrix}}f_{X,Y}(x,y) \ \ \ 단, z=g(x,y) , w=h(x,y)\]
여기서 분모 부분은 야코비안이며 다음과 같이 정의된다.
\[\frac{\partial (z,w)}{\partial(x,y)} \equiv det \begin{pmatrix} \frac{\partial z}{\partial x} & \frac{\partial z}{\partial y} \\ \frac{\partial w}{\partial x} & \frac{\partial w}{\partial y}\end{pmatrix}\]
ex)
Q. X,Y의 결합분포의 확률밀도 함수를 \(f_{X,Y}(x,y)\)라 하고, \(Z \equiv 2X e^{X-Y}\)와 \(W \equiv X-Y\)의 결합분포의 확률밀도함수를 \(f_{Z,W}(z,w)\)라 할때, \(f_{Z,W}(6,0)\)을 \(f_{X,Y}\)로 나타내라.
A.
먼저, X와 Y는 다음과 같이 나타낼 수 있다.
\[X=\frac{Z}{2e^{-W}} , Y=\frac{W}{2e^{-W}}-W\]
이제 야코비안을 구한다.
\[\begin{pmatrix} Z \\ W\end{pmatrix} = \begin{pmatrix} \frac{\partial z}{\partial x} & \frac{\partial z}{\partial y} \\ \frac{\partial w}{\partial x} & \frac{\partial w}{\partial y}\end{pmatrix} \begin{pmatrix} X \\Y \end{pmatrix} \\
\Rightarrow \begin{pmatrix} Z \\ W\end{pmatrix} = \begin{pmatrix} 2(e^{x-y}+xe^{x-y}) & -2xe^{x-y} \\1 & -1\end{pmatrix} \begin{pmatrix} X \\Y \end{pmatrix}\]
그 다음 야코비안의 determinant를 구한다.
\[\begin{vmatrix} 2(e^{x-y}+xe^{x-y}) & -2xe^{x-y} \\1 & -1 \end{vmatrix} = -2e^{x-y}-2xe^{x-y} + 2xe^{x-y} = -2e^{x-y}\]
이제 이를 식에 대입하면
\[f_{Z,W} (z,w) = \frac{1}{\begin{vmatrix}-2e^{x-y} \end{vmatrix}}f_{X,Y}(\frac{z}{2e^{-w}},\frac{z}{2e^{-w}}-w)\]
마지막으로, z=6,w=0일 때, x와 y는 각각 3,3 이다. 따라서 식은 다음과 같이 완성된다.
\[f_{Z,W}(6,0)=\frac{1}{\begin{vmatrix}-2e^{3-3}\end{vmatrix}}f_{X,Y}(3,3)=\frac{1}{2} f_{X,Y}(3,3)\]
확률밀도의 기댓값, 분산, 표준편차 예제
ex1)
Q1. 확률변수 \(X\)의 확률밀도함수 f_{X}(x)가 다음 식으로 주어질 때 \(E[X]\)와 \(E[X^2]\)의 값
\[f_{X}(x) = \begin{cases}2x \ (0 \le x \le 1) \\ 0 \ \ \ (기타) \end{cases}\]
A1.
\(E[X] = \int^{\infty}_{-\infty} xf_{X}(x)dx = \int^{1}_{0}x(2x)dx = \left[\frac{2}{3}x^{3} \right]^{1}_{0} = \frac{2}{3} \\
E[X^2] = E[g(X)] = \int^{\infty}_{-\infty} g(x)f_{X}(x)dx = \int^{1}_{0}x^2(2x)dx = \left[ \frac{2}{4}x^{4} \right]^{1}_{0} = \frac{1}{2}\)
Q2. 다음의 성질을 적분 계산으로부터 유도하라
- \(E[3X] = 3E[X]\).
- \(E[X+3] = E[X]+3\).
A2.
\[E[3X]=\int^{\infty}_{-\infty}3xf_{X}(x)dx =3\int^{\infty}_{-\infty}xf_{X}(x)dx=3E[X] \\
E[X+3]=\int^{\infty}_{-\infty}(x+3)f_{X}(x)dx = \int^{\infty}_{-\infty}xf_{X}(x)dx+3\int^{\infty}_{-\infty}f_{X}(dx)dx = E[X]+3\times1=E[X]+3\]
Q3. 확률변수 \(X\)의 확률밀도함수 \(f_{X}(x)\)가 다음 식으로 주어질 때 분산 \(V[X]\)와 표준편차 \(\sigma\)를 구하시오.
\[f_{X}(x)= \begin{cases}2x \ (0 \le x \le 1) \\ 0 \ \ \ (기타) \end{cases}\]
A3.
위에서 \(E[X]=2/3\)을 구했으므로
\[V[X]=E \left[ \left(X-\frac{2}{3} \right)^2\right] = \int^{\infty}_{-\infty} \left(x-\frac{2}{3} \right)^{2} f_{X}(x)dx = \int^{1}_{0} \left(x-\frac{2}{3} \right)^{2} (2x)dx \\
\int^{1}_{0} \left( 2x^{3} - \frac{8}{3}x^{2} + \frac{8}{9}x \right)dx = \left[\frac{1}{2}x^{4}-\frac{8}{9}x^{3}+\frac{4}{9}x^{2} \right]^{1}_{0} = \frac{1}{2}-\frac{8}{9}+\frac{4}{9} = \frac{1}{18} \\\]
혹은 분산의 성질에 의해
\[V[X]=E[(X- \mu)^2] \\ = E[X^{2}-2X \mu + \mu^2] \\ = E[X^2]-2E[X]E[X] + E[X]^2 \\ = E[X^2]-E[X]^2\]
이므로
\(V[X]=E[X^2]-E[X]^2 = \frac{1}{2} - \frac{4}{9} = \frac{1}{18}\)
로 구할 수 있다.
\[\sigma =\sqrt{V[X]} = \sqrt{\frac{1}{18}}=\frac{1}{3 \sqrt{2}}\]
21 Jul 2020
•
Backbone
ResNet은 2015 ILSVRC, COCO 대회에서 우승한 모델이며 특히 3.6%의 top-5 error를 달성함으로써 사람의 분류 기준인 5% 내외를 뛰어 넘었다.
More …
17 Jul 2020
•
Statistics
확률 0
다음과 같은 확률변수 X를 생각하자
- \(\omega = (u,v) , \ 0 \le u \le 1 , \ 0 \le v \le 1\).
- \(X(\omega)=10u\).
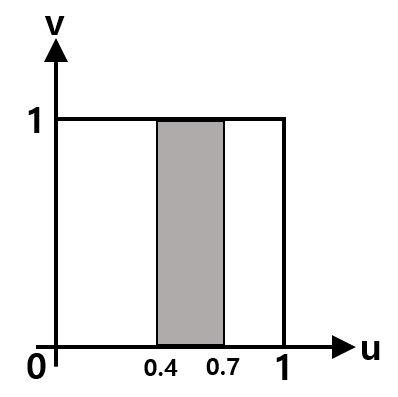
이 경우 \(P(4 \le X \le 7)\) 확률은 아래 그림과 같이 사각형의 면적을 구하면 될 것이다.
하지만 만약 \(P(X=2)\) 확률을 구하고자 한다면 다음과 같이 구해야 한다.
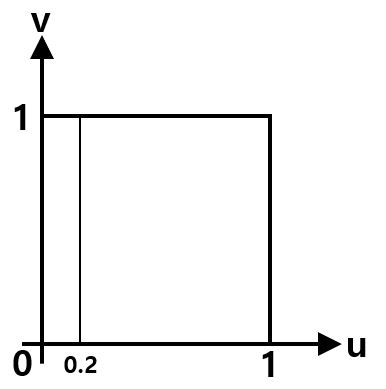
그러면 이 경우에는 \(P(X=2)=0\)이라는 결과가 얻어진다.
이 예제로 연속된 실수값에서는 특정한 확률의 값은 0이 된다는 것을 알 수 있다.
확률 밀도 함수
연속된 실수값에서 확률을 구하기 위해서는 누적분포함수와 확률밀도함수를 사용한다.
여기서 누적분포함수는 ‘X이하의 값이 나올 확률’ 정도로 기억하면 편하다.
\[F_{X}(a) \equiv P(X \le a)\]
확률밀도함수는 말 그대로 확률의 농도라고 해석할 수 있다. 즉, 확률밀도함수의 값이 크다는 것은 그 부근의 확률이 높다는 것을 의미한다. 식은 아래와 같이 누적분포함수의 변화량으로 구할 수 있다.
\[f_{X}(x) \equiv F'_{X}(x)= \frac{dF_{X}(x)}{dx}\]
위에서 말한것과 같이 연속값의 확률변수의 순간적인 확률은 정의되지 않는다. 확률변수의 특정한 범위에 대해 그 범위가 나올 확률은 다음과 같이 정의된다.
\[\int_{\beta}^{\alpha}f_{X}(x)dx\]
물론 확률의 총합은 1이기 때문에 다음이 성립한다.
\[\int_{-\infty}^{\infty}f_{X}(x)dx=1\]
균등 분포
확률밀도함수로 나타내는 확률분포가 구간 \([\alpha,\beta]\) 위의 균등분포일때 식은 다음과 같이 정의된다.
\[f_{X}(x)= \begin{cases} \frac{1}{\beta - \alpha} \ (\alpha \le x \le \beta) \\ 0 \ (기타) \end{cases}\]
확률밀도함수의 변수 변환
\(X\)의 확률밀도함수 \(f_{X}\)가 주어져 있을 때, \(Y=3X-5\)라고 할 때 \(Y\)의 확률밀도함수 \(f_{Y}\)를 구해보자.
단순히 생각했을 때는 \(f_{Y}(4) = f_{X}(3)\)이 될 것 같지만 두 확률밀도함수는 서로 선형관계에 있지않다. 자세히 생각해보면 만약 \(f_{X}\)가 범위 \(0 \le X \le 3\)에서 1의 확률을 가진다면 Y는 \(-5 \le Y \le 4\)에서 확률이 1이 될것이다. 오히려 범위가 3배나 증가했고 변화량은 이에 분명히 반비례할 것이다. 변화량이 반비례 하다는 것은 결국 확률밀도 함수는 다음과같이 정의 된다는 것이다.
\[f_{Y}(y) = \begin{vmatrix} \frac{f_{X}(x)}{g'(x)} \end{vmatrix}\]








